發布時間:2022-03-06所屬分類:計算機職稱論文瀏覽:1次
摘 要: 摘要 在過去的10年中, 以基因組學、醫學遺傳學和神經信息學等為代表的生命科學各研究領域, 以前所未有的增長趨勢, 積累了海量的數據信息. 這些數據類型復雜、數量龐大, 其中蘊含的價值更是不可估量. 通過傳統的處理手段, 難以理清海量原始數據中錯綜復雜的關聯信息. 而
摘要 在過去的10年中, 以基因組學、醫學遺傳學和神經信息學等為代表的生命科學各研究領域, 以前所未有的增長趨勢, 積累了海量的數據信息. 這些數據類型復雜、數量龐大, 其中蘊含的價值更是不可估量. 通過傳統的處理手段, 難以理清海量原始數據中錯綜復雜的關聯信息. 而針對生物大數據的可視化研究, 將有利于科研人員對復雜數據進行多角度觀察并獲取有效信息. 生物數據量越大, 復雜性越高, 可視化在生物有效信息挖掘方面發揮的作用就越大. 本文通過例舉若干生物機構中心現存的數據規模和數據增長速率, 說明生物研究領域已進入大數據時代, 然后由生物數據的組成特征及可視化的特點引出生物大數據可視化的重要性和必要性. 本文總結了生命科學研究領域中不同類型生物大數據的可視化研究進展, 最后討論了目前生物大數據可視化所面臨的挑戰, 并提出可能的解決方案.
關鍵詞 大數據生物信息學可視化
2005年以來, 隨著高通量測序技術的不斷進步及廣泛應用, 生命科學跨入了大數據時代. 以基因組科學和生物醫學領域為代表的生命科學研究產生和積累了海量的數據信息: 歐洲生物信息學研究所 (European Bioinformatics Institute, EBI)目前存儲了將近20 PB的數據, 其中基因組數據約占2 PB, 這一數字隨著新一代測序技術的不斷發展每年成倍增長[1]; 高通量測序數據庫(Sequence Read Archive, SRA)作為美國國立生物技術信息中心(National Center for Biotechnology Information, NCBI)最主要的高通量數據存儲庫, 目前存儲的數據總量超過了3 PB, 對外發布的數據量達到1640 TB[2]; 此外, 當前世界上最大的基因數據產出機構——華大基因研究院(Beijing Genomics Institute, BGI)每天產出包括人、植物、動物和微生物在內的約6 TB基因組數據[1].
國際上的多個合作研究項目產生了史無前例規模的生物數據. 為了破譯人類的全部遺傳信息, 美國科學家在1985年率先提出了人類基因組計劃(Human Genome Project, HGP)[3], 這一計劃不僅覆蓋了 99.99%的人類基因組, 解讀了人體基因密碼的“生命之書”, 而且推動了生命科學和生物技術的基礎性研究, 促進了一系列科學技術的產生和發展; 2004年, 為了尋求新一代DNA研究技術對人類基因調控序列在全基因組水平上研究的應用, “DNA元件百科全書” 計劃(Encyclopedia of DNA Elements, ENCODE)啟動, 這一計劃促使來自32個科研機構的442名研究人員獲取并分析了超過15 TB的原始數據[4]; 從2005年底至今, 由美國國國家癌癥研究所(National Cancer Institute, NCI)和美國國家人類基因組研究所(National Human Genome Research Institute, NHGRI)共同發起的癌癥基因組圖譜計劃(The Cancer Genome Atlas, TCGA)[5], 通過多個基因組技術平臺分析并獲取超過 800 TB數據及文檔資料, 為診斷、治療和預防癌癥打下了敦實的基礎; 2010年, 中、英、德、美等國共同合作了國際千人基因組計劃(1000 Genomes Project), 至今為止產生的數據量達到50 TB, 其中包含來自全球27個族群的2500個人的全部基因組信息[6].
當今測序技術的進步速度之快, 已遠超計算機領域里的摩爾定律[7](價格不變時, 集成電路的性能每18個月增加一倍). 在1990年啟動的人類基因組計劃中, 美、歐、中、日等多個國家和地區超過200名科學家, 投入了超過10年的時間和約30億美元才完成人類全基因組的測序; 但現在, 僅靠一個實驗室的數名研究人員, 就可在數周內完成人類全基因組重測序, 而試劑成本則可控制在1000美元之內. 如此巨大的進步, 不僅給生命科學的研究帶來了巨大的機遇, 在此基礎上如何有效處理和分析這些測序數據, 也給此領域內的研究人員帶來了巨大的挑戰.
以DNA序列數據為代表的海量數據是構成生命科學研究的重要組成部分, 通過應用生物信息學技術進行大數據研究, 理解隱藏在大數據里的生物學知識成為當前生物技術發展的迫切需求. 傳統的基于文本的數據處理和展示模式已經嚴重制約了對于生命科學大數據的解讀. 基于可視化技術的信息挖掘成為一種必需的解決途徑. 可視化是對事物建立心理模型或者心理圖像的一個過程1). 通過可視化, 抽象的符號信息可以轉化為易于理解的圖像和模型, 另外交互式的使用允許研究人員從不同的可視化角度來探究隱藏在大數據里的不同模式和關聯. 可視化擁有強大的將復雜數據轉化為可利用信息的能力. 生物數據復雜、冗余等諸多特點決定了可視化是有效地理解生物數據的不可或缺的手段. 生物大數據可視化依托于現有的計算技術, 在一定時間內產生視覺表現模型, 并在此基礎上盡可能地增強交互性, 從而加強用戶體驗以及對生物數據分析結果的認知能力.
1 生物大數據的特征及來源
生物大數據除了具有傳統大數據4“V”的特點, 即數據量大(Volume)、數據處理速度快(Velocity)、數據源多變(Variety)和蘊含價值(Value)外[8], 還擁有其特有的數據復雜性(Complexity)[9]. 有生物學家提出, 復雜程度將生物領域產生的大規模數據與其他科學領域的產出區分開來. 在高能物理中, 數據有著合理的結構和注釋, 而生物學數據目前來講卻難以完美地組織起來. 除了簡單的基因組測序外, 生物學家會追蹤許多不同的細胞和分子成分, 試圖使用各種手段弄清其中包含的復雜關系. 此外, 由于生物數據經常來自不同的實驗方法和機構, 使用了不盡相同的參數標準, 產生的數據類型豐富多樣, 導致這些數據可能采用不同的存儲結構(如narrowPeak, BED, SAM 等), 針對不同的研究對象(如基因序列、蛋白質互作關系、菌群共生等), 來源于不同的渠道(如測序、醫療記錄等). 不同實驗的參數標準、特異的細胞組織類型以及無法結構化存儲的藥物處理過程等諸多因素都是造成生物大數據復雜性的原因. 生物數據可視化的核心就是利用有效的算法消除這些數據的復雜性, 從而將其中隱含的生物學規律清晰地展示給用戶, 而解析、轉換這些復雜數據的格式, 則是數據可視化設計的第一步, 下面以數據來源為分類來了解生物數據復雜多樣的格式特征.
首先, 測序技術的飛速發展為生物領域提供了數目龐大的寶貴資源. 目前第二代測序技術被廣泛采用, 第二代測序產生數以百萬計的短序列, 再由拼接算法將這些短序列在全基因組范圍內組裝起來, 從而進行進一步的數據分析工作[10]. 迄今為止, 新興的單細胞測序技術一直被認為是最為值得關注的測序技術, 傳統的測序方法忽略了細胞間的差異性, 得到的結果僅僅是一群細胞信號的平均值, 而基于單細胞水平對全基因組進行擴增與測序的單細胞測序技術, 不僅在基因表達量方面測量精準, 而且能夠檢測到表達量較低的基因及非編碼RNA, 因此具有很大的優勢及發展空間[11]. 除此之外, 單細胞 RNA測序(single-cell RNA-seq)使追蹤單個細胞的轉錄組成為可能 [12], 染色質免疫共沉淀測序 (ChIP-seq)[13]等實驗技術有力地支持了對基因組數據的功能性注釋. 這些高通量的測序技術, 為研究者發現與疾病相關的基因型變異、研究某個表型的整個轉錄組、某一條件下的甲基化狀態以及對DNA上蛋白質結合位點進行定位等工作提供了便利與支持, 然而隨著數據規模的增大, 測序數據的處理和分析逐漸成為瓶頸.
其次, 生物芯片技術的使用在過去的數年中產生了龐大的數據資源. 為了實現對生物組織、細胞、蛋白質、核酸等組分中富含的大量信息進行快速準確的檢測, 研究人員在固體芯片表面構建了微型的生物化學分析系統. 當前的生物芯片主要分為微陣列芯片和微流控芯片兩種類型[14]. 傳統的以靜態和雜交技術為基礎的微陣列芯片主要有基因芯片(DNA Microarray)、蛋白芯片(Protein Chip)和芯片實驗室 (Lab-on-a-chip)等形式[15]. 其中, 基因芯片也叫DNA 高密度微點陣雜交技術, 以核酸探針互補雜交技術為基礎而建立, 可用于DNA序列測序、基因表達分析、基因分型以及基因多態性分析等研究目的; 蛋白芯片依據蛋白質分子和其他分子的相互作用而構建; 而芯片實驗室將整個流程集約化形成微型的分析系統. 芯片與生物分子反應所產生的信號需要借助于芯片掃描儀, 并通過相關軟件分析采集到的各反應點的熒光強弱信號、所在位置信息所形成的圖像來獲取有關的生物信息. 微流控芯片以微流體控制技術為基礎, 主要有毛細管電泳芯片、PCR反應芯片等形式[15]. 近年來, 生物芯片技術在基因表達水平檢測、基因診斷、藥物篩選、個體化醫療臨床、疾病診斷和治療、疾病易感基因發現以及基因功能確認等醫學與生物學領域得到廣泛的應用.
再次, 生物質譜為生命科學的研究做出了巨大的貢獻, 不僅被認為是大規模、高通量鑒定幾十萬分子量的生物大分子結構的首選工具, 而且對于研究蛋白-蛋白等大分子之間的相互作用、翻譯后修飾以及基因表達水平的變化有著很大的幫助. 質譜法主要原理是先將樣品變為氣態的離子混合物, 再按照質荷比(m/z)進行分離, 從而成功獲取樣品的質量、含量及結構等信息[16]. 在獲取使用譜圖法或列表法表示的測定結果后, 需要進行進一步的數據分析. 對于鑒定蛋白質的方法, 目前常用的有質量紋鑒定法 (Peptide Mass Fingerprinting)、二級質譜的數據庫搜索鑒定法(MS/MS Database Searching)等手段[17]. 質譜分析技術被稱作蛋白質組的核心技術, 最近在 Nature上公布的人類蛋白質組草圖就是基于16857個質譜分析實驗結果的整合[18]. 基質輔助激光解析-飛行時間質譜系統(VITEK®MS)作為美國FDA批準的首個用于檢測病菌的質譜檢測系統, 可用于酵母菌和致病細菌臨床快速鑒定, 這也是第一種能在數分鐘內檢測致病微生物的醫療器械[19].
此外, 通過各種先進手段獲取的與生物相關的圖片影像資料也日益豐富起來. 生命體內存在著蛋白質、RNA以及DNA等種類繁多的生物大分子. 隨著顯微鏡、成像捕捉等高精尖端儀器技術的不斷發展, 科學家們不僅能夠通過低溫電子顯微鏡直接觀察到蛋白質等生物大分子精細到原子的組織結構, 而且逐漸可以直接觀測記錄到活體組織中生物大分子在時間、空間維度上的結構變化和各分子間的相互作用的動態畫面. 目前, 美國斯坦福大學研究人員借用 “微型內窺鏡”及玻璃導管已經實現了在不破壞活體被觀察組織的情況下, 長時間地對活體大腦神經元進行觀測[20]; 北京大學開發的“生物正交受激拉曼散射成像”技術成功地特異性標記了活細胞的脂類、核糖、蛋白質和糖類等成分[21]; 美國紐約冷泉港實驗室將分子標記手段與顯微鏡技術相結合, 順利完成了第一個活體老鼠體內腫瘤細胞活動的影像記錄工作[22]. 通過這些高新技術手段, 科學家們有望從中得到所有細胞、組織中蛋白質和復合物的相關位置, 弄清人體的有機物概況. 因此, 越來越多非結構化的圖片影像數據亟待批量化整合、分析及展示.
最后, 臨床數據也是一個不可忽略的數據來源. 僅隸屬于中國中醫科學院的廣安門醫院每年產生的數據量高達就70 TB2), 如果將全國的臨床數據都集合在一起, 其數據規模更是不可估量. 現有的臨床醫學數據包含電子病歷、醫學影像、化驗結果以及生化檢查、病理切片檢查的生物學信息等, 這些臨床信息不但多樣、冗余、不完整, 而且往往涉及患者隱私、公司利益沖突等問題, 加之有些數據之間難以關聯, 造成標準化實施的困難. 這種結構化與非結構化格式并存的特點, 使得臨床數據的整理分析變得異常困難[23]. 為了挖掘這些醫療數據中潛在的價值, 一些臨床和科研機構著手將醫療數據進行整合, 構建臨床試驗數據的共享和分析平臺. 北京的各大醫院通過臨床科研信息共享系統將實踐數據化、規范化、數字化, 海量的數據通過整理轉換等過程, 被進一步應用在查詢檢索、統計分析和數據挖掘上, 以此獲取新的知識, 從而更加有效地對臨床實踐進行指導2). 美國臨床腫瘤學會(American Society of ClinicalOncology, ASCO)旗下的“CancerLinQ”允許研究人員進入、訪問和分析匿名癌癥患者的病例[24]; 新型的電子診斷領域也為信息整合提供了極大的便利. 海量的臨床數據的整合利用將大大有助于科研人員及醫學專家對大規模疾病患者群體治療情況進行分析, 從而為攻克疑難雜癥提供契機.
相關知識推薦:收數據可視化論文的期刊
除了上述幾個主要的生物大數據來源以外, 新型的技術手段不斷貢獻出寶貴的資源數據, 例如最新的流式熒光技術[25]可以實現快速、準確、高通量地對腫瘤標志物進行檢測, 此外不同類型的儀表設備也為生物領域提供了不少有價值的數據. 豐富的數據來源顯示出生物數據不僅數據規模龐大, 類型復雜多變, 而且在立體空間上結構、位置隨時間不斷變換、移動. 解決這些數據的存儲只是最基本的任務, 更為重要的是使用這些數據. 同樣, 對生物大數據進行可視化是為了更加充分地挖掘出數據中潛在的價值, 因此在設計可視化工具時如果能夠以數據來源為依據, 從數據規模、復雜度、空間性和時間變換性這4個方面針對目標數據進行考慮, 將十分有益于從數據中獲取有效信息.
2 生物大數據可視化類型及現狀
可視化對生物數據的分析至關重要, 以生物數據的特性來看, 一般情況下僅憑文字很難描述清楚其中存在的復雜關系. 可視化不僅可以用來進行形象展示, 更是數據分析的第一個戰場, 對生物數據進行良好的直觀、交互性展示可以揭示出數據內在的錯綜復雜的關聯狀況, 在這一點上其他方法很難與可視化相提并論. 從最簡單的Excel電子表格、Google 文檔到R, Pandas等統計編程架構, 再到D3.js, Prefuse 等可視化程序包, 這些通用數據可視化和處理工具都可以為數據分析、信息挖掘提供很好的計算機手段. 另外針對于不同的數據類型和目的, 生物領域涌現了一大批開源、優秀的可視化工具(圖1), 這些針對生物研究人員開發的工具易于上手, 為生物數據的快速分析提供了便利.
2.1 測序數據
測序技術、生物芯片提供了存在于生命體中的 DNA, RNA, 蛋白質等大分子的豐富的一級序列資源, 現有的基因組瀏覽器根據不同的需求對這些基序進行了從細節到宏觀的展示. 以當前最為常用的 UCSC Genome Browser[26]為例, 它支持可以被比對到基因組上的任何數據類型, 將圖像在服務器端渲染后嵌入網頁中. 它對于基因組數據的展示模式體現了大多現存瀏覽器共有的特點: (1) 以染色體位置為索引的基因組數據視圖; (2) 以參考基因組為標準提供位置坐標軸; (3) 基于track展示; (4) 良好的交互性和可定制性, 可根據用戶所需進行裝載或隱藏數據內容. 除了這些展示特征外, 不同的基因組瀏覽器也擁有自己獨特的功能. GenomeView[27]提供注釋編輯器, 可以展示和注釋信息, 進行多序列比對、共線性匹配、短序列比對以及其他可以被顯示的內容; 交互探究大型集成數據集的可視化工具 (Integrative Genomics Viewer, IGV)[28]可支持多種數據類型的交互展示, 包括測序序列比對、基因表達數據和拷貝數異常(圖1(e))等.
由于不同的組織轉錄組的表達差異往往借助于統計手段進行聚類, 并需要使用熱圖使聚類結果呈現直觀的展示, 并加以解釋, 聚類得到的不同表達模式還可進一步按照功能富集程度進行分類并以圖形化方式表示假設檢驗的結果, 以Gitools[29]為代表的此類工具采用了熱圖的形式對基因組數據進行集成化分析和展示, 此工具通過引入KEGG, Biomart等生物數據庫達到對先驗知識的利用, 提供富集分析、相關性分析以及顯著性計算等豐富的分析手段, 通過集成排序、過濾、移動、聚集、搜索及可視化行列注釋等功能允許使用者交互性地分析和可視化多維數據.
此外, 測序數據的可視化可能會對數據的深入挖掘起著決定性的作用. 例如, 單核苷酸多態性 (SNP)、插入缺失標記(InDel)以及基因組結構變異是一級序列中頗受關注的內容, 它們往往與復雜疾病的發生發展有著密切關系. 其中, 基因組結構變異包括插入、刪除、倒置、易位、復制以及拷貝數變異等不同的類型, 每種類型使基因組產生不同的結構改變. 由于各類結構變異的復雜性, 以及真核生物基因組結構固有的重復序列特性, 導致僅憑現有的算法很難完全正確地檢測出每種類型的變異. 特別地, 結構變異往往會引起短序列的錯誤定位, 進而導致小尺度的多態性預測錯誤, 因此通過提供可視化工具來方便研究者進行人工判斷在結構變異的檢測和識別中變得不可或缺[30]. 目前已有諸多的致力于展示、探究結構變異的可視化工具, 如可以運行在各種操作系統上的針對結構變異的集成軟件inGAP-sv[30], 不僅能夠以較低的假陽性概率檢測出復雜的變異類型, 而且提供了友好的可視化接口, 每種類型結構變異特征模式進行標識, 通過右擊鼠標可獲取關于特定讀長或結構變異的所有信息(圖1(f)). 除此之外, inGAP-sv允許使用者根據自身的需求靈活設定顯示測序短序列的外型和連線的顏色, 以便更好地為探究結構變異提供便利. inGAP-sv針對于結構變異提供識別、可視化、注釋、人工編輯等一站式的服務, 這種集可視化、挖掘為一體, 注重用戶體驗度的工具設計方式預示了未來的軟件開發走向.
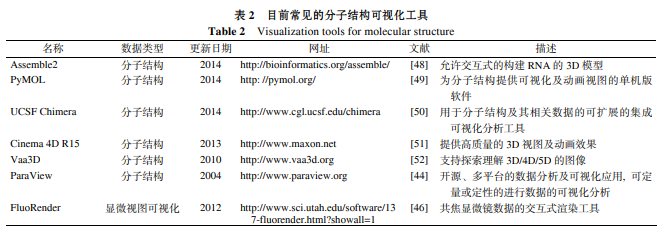
2.2 分子結構數據
結構分子生物學是將物理和化學與生物學相連接的一門關鍵學科, 它主要聚焦于3D和4D復雜形狀和功能關系的研究, 熒光標記、顯微觀察以及成像捕捉等技術為這一領域提供了豐富的視圖數據, 而眾多服務于分子結構的可視化工具在研究過程中起到了極大的作用. 以可視化軟件ParaView[44]為例, 它允許使用者通過定性和定量的技術手段對大量的數據集快速建立3D視圖模型, 從任意的角度對分子結構進行觀察. 由于蛋白質等大分子結構精細復雜, 其內部的位置關系需要大量的計算資源, 因此3D視圖軟件往往比2D展示工具需要更加高效的算法設計, 高性能的計算設備以及高分辨率的展示屏幕. 為了增強對大規模數據集的處理能力, ParaView使用了分布式存儲計算資源, 可以運行在超級計算機上來對萬億次級的數據集進行可視化分析. 除了ParaView, Amira[45], FluoRender[46]等工具都可以用來瀏覽分析 CT, MRI和顯微圖像, 以及實現對分子結構的3D 還原.
這些以計算圖形學為基礎所開發的軟件工具雖然以更為精細準確的展示方式取代了物理模型, 但是卻失去了與物理對象互動接觸時產生的固有的視覺豐富性, 而這種觸覺和本體感受往往為理解3D模型和進行物理操作提供了關鍵的線索. 因此工業領域的物體分層制造逐漸被應用在對分子結構的還原上. 譬如最近清華大學與美國德雷克賽爾大學研究人員以混合膠、纖維蛋白和宮頸癌細胞為原材料, 在精準的參數控制下, 利用一臺3D細胞打印機成功制造出了與自然腫瘤十分接近的腫瘤模型[47].
2.3 關系網絡
生物領域中由于生物分子互作、代謝途徑、調控作用和基因表達等現象的存在促使了各種各樣的關系網絡的存在, 隨著科學家們對這些過程的深入研究, 人們對其復雜度的了解也在不斷增加. 生物學家經常需要對此類有相互作用關系的復雜系統和高維數據進行分析, 因此產生了可以對各種網絡關系進行可視化的軟件工具. 目前常用的復雜網絡可視化工具有Cytoscape[53]、R中的igraph包以及Perl中的 GraphViz包等. Cytoscape代表一類以點線模式為基礎進行網絡可視化的工具, 它提供基礎的功能布局和網絡查詢功能, 并且能夠依據基本數據關系動態生成可視化網絡. 其中因子、蛋白質和分子使用點表示, 兩點間的交互關系用連接也就是邊進行表示. 這種表示模式整合了分子間相互作用的網絡, 適用任何分子系統的結構和相互關系, 允許將蛋白質、DNA 和其他對人類和生物有重要作用的分子數據庫關聯起來, 形成龐大的網絡結構. 此外, R中的NetBioV, Gephi[54](圖1(c))等軟件包為生物信息學者提供了對節點連接類型的網絡關系可視化開發工具.
隨著計算手段的進一步發展, 網絡關系的3D可視化形式逐漸發展起來. BioLayout Express3D[55]可以用于在2D, 3D空間內的可視化、分類歸納、探索和分析大型的網絡關系. 此軟件可對蛋白質互作和序列相似性等關系形成的網絡進行展示, 摒棄了傳統的對微陣列基因表達數據進行統計學差異分析的方法, 轉而基于關聯度評估來定義表達量間的相似性, 從而形成數據分析的網絡范式 , 而且此工具基于 OpenCL并行框架編寫, 充分考慮到網絡關系3D可視化時所需的計算資源及圖形處理技術支持等問題. 在2D或3D環境中BioLayout Express3D提供以下3個功能; (1) 對圖像的移動、翻轉和縮放操作; (2) 節點、邊的個性化定制, 且允許設定文本標簽以加強示意功能; (3) 背景顏色、3D燈光和投影、節點表面紋理等顯示內容可進行偏好設置, 以便更好地對可視化效果進行渲染.
2.4 臨床數據
雖然電子病歷的使用范圍在不斷地擴大, 但是不統一的標準、非結構化的數據模式對研究者獲取疾病治療的真實資料造成了很大的障礙. 科學家們也開始著手處理這個問題, 以整合人類腫瘤數據為目標的Flatiron就是其中一個代表, Flatiron搭建的基于云端的OncologyCloud[63]平臺聚合并轉換了來自多渠道的患者信息、藥單信息和患者恢復狀況等數據, 并提供對數據集的歸納分析(圖1(d)), 由此醫生不僅能夠通過OncologyCloud看到同類患者的治療結果, 還能追蹤到以往不同治療方案所產生的臨床結果. 這樣一個提供全面的腫瘤數據收集、分析的系統也為腫瘤領域的基礎研究提供了極大的便利; “癌癥生命科學協會CEO圓桌會(the CEO Roundtable on Cancer)” 推出的PDS計劃(Project Data Sphere)[24], 嘗試打造一個癌癥三期臨床試驗數據共享和分析平臺, 數據集由賽諾菲、輝瑞以及阿斯利康等機構共同提供, 這些數據集在去除患者個人信息后進行了統一編號. 由于舊習慣及某些規章制度的影響, 大量醫療數據的整合和挖掘還需時間來逐步發展和規范. 但不可否認的是, 將治療信息匯集在一起進行分析展示對攻克疾病有著不可忽視的作用.
除了以上闡述的可視化工具, 根據不同的需求還存在著很多其他的可視化形式 (表 1~4). 例 如 , Chimera[50](圖1(b))將分子結構和包括密度圖譜、超分子裝配、序列比對、軌跡在內的相關數據集成起來, 產生高質量的動畫效果; 由于不同質譜儀所產生的蛋白質譜初始數據格式不同, 而蛋白質組學質譜數據分析中統計學算法的實現過于復雜, 數據表示可視化、特征提取可視化及分類可視化對蛋白質質譜數據的分析十分重要; 除此之外, 還存在針對于SNP展示、表觀遺傳學所提供的核小體定位及組蛋白分析結果的可視化、微生物群落概況的可視化分析、海藻圖解等諸多專項專能的可視化軟件工具. 生物大數據可視化工具種類繁多, 為了更好地為挖掘有效信息做鋪墊, 其開發趨勢向具有統計分析功能的一站式集成工具靠攏. 此外, 未來的生物大數據可視化工具在交互性、美觀性、實用性方面會做得越來越好.
3 展望及未來的挑戰
生物數據有著自己的特點, 不僅數據規模龐大, 分布在不同的組織機構, 而且維度高, 數據不完整性和不確定性強. 利用各種技術手段獲取數據本身不是目的, 將數據進行可視化也不是目的, 真正的目的是探究生命的本質, 發現未知的規律, 為人類的健康幸福服務, 因此挖掘隱藏在數據背后的涵義成為生物信息學家們一致的目標. 充分了解目前在分析生物數據的道路上存在的一些挑戰及潛在的解決方案具有重要的意義.
首先, 現有的海量生物數據中存在著大量的冗余和噪音, 生產數據的組織機構可以對原始數據進行標準化處理和質控. 例如, 可將數據分門別類, 使用統一的數據存儲標準、規格等. 合理的預處理手段可在一定程度上降低數據規模及復雜度, 節省存儲空間及數據傳輸成本, 同時也會提高數據的易讀性, 減少研究者對數據進行相同處理所需要的計算時間和資源等.
其次, 由于產出的數據往往分布在不同的研究機構, 如何實現海量數據的共享是研究人員們普遍面臨的一大挑戰. 現有的分布式注釋系統(DAS)[69]提供了一個潛在的解決方案. 它定義了一份用來交換基因或蛋白質序列及其注釋的通信協議, 在此協議下, 基于網絡的可視化系統可實現同一界面下對遠程異地分布注釋數據的可視化.
再者, 生物數據特有的復雜多樣性給數據挖掘帶來很大困難, 因此在對大批量數據進行可視化前, 數據投影及各種降低維度的技術被廣泛采用. 與此同時, 人類視覺的敏銳性、使用者面對展示界面時的推斷能力和信息搜索能力等因素都需要加以考慮. 對生物大數據進行可視化時, 需要記住目標使用者是人, 目的是信息的展示和探索, 而非一味地追求視覺美觀. 在開發生物大數據的可視化工具時, 需要盡可能提高軟件或平臺的易用性, 充分考慮用戶的體驗度, 提供友好的交互界面.
此外, 在有限的時間內對大規模數據進行處理及可視化是最基本的要求. 除了通過使用優化算法對數據規模和可視化效率進行平衡外, 還可以引入并行處理技術. 在對若干數據集進行可視化時, 可將查詢處理分散在多個并行節點上, 以此縮短運行時間, 加快可視化的速度.
除了前述內容, 用于傳輸生物數據的網絡基礎設施的建設、數據的存儲方式等諸多方面都存在著一定的困難. 雖然在分析生物大數據的道路上面臨著諸多挑戰, 但是這些暫時的困難并不能阻止科學家們前進的腳步, 生命科學的神秘面紗最終將會在一代代科研人員的努力下被完全揭開.——論文作者:周琳① , 孔雷② , 趙方慶①*
SCISSCIAHCI